文献追踪 |突破壁垒!scCUT&Tag 单细胞染色质修饰测序新探究

一
接下来,让我们一同深入探究这篇论文,了解scCUT&Tag技术是如何构建的,它在细胞分化和肿瘤研究中取得了哪些重要成果,以及该技术为未来生命科学研究开辟了怎样的新路径。

Nature Biotechnology ( IF 33.1 ) Pub Date : 2021-04-12 , DOI: 10.1038/s41587-021-00865-z
二
-
研究思路:

-
文章亮点:
三
实验设计与方法:研究人员对 H1 人胚胎干细胞(hESCs)进行 CUT&Tag 实验,使用抗 H3K27me3 特异性抗体进行批量处理,之后在基于纳米孔的 ICELL8 系统上对单细胞进行聚合酶链反应(PCR)和文库富集(图 1a)。同时,将实验结果与之前发表的 K562 细胞和 hESCs 的 H3K27me3 scCUT&Tag 数据进行比较。
实验结果:经过质量控制筛选后,通过统一流形逼近和投影(UMAP)嵌入分析发现,804 个 hESCs(中位数为 375 个独特片段)和 908 个 K562 细胞(中位数为 6,064 个独特片段)能被清晰分开(图 1b)。hESCs 的独特片段数量仅为 K562 细胞的 6%,这表明干细胞的整体 H3K27me3 水平低于分化程度更高的细胞类型。即便将每个细胞的独特片段数量下采样至相同中位数,H3K27me3 信号依然能够区分这两种细胞类型,这充分证实了聚类分析是由 H3K27me3 信号差异驱动,而非独特片段数量差异。
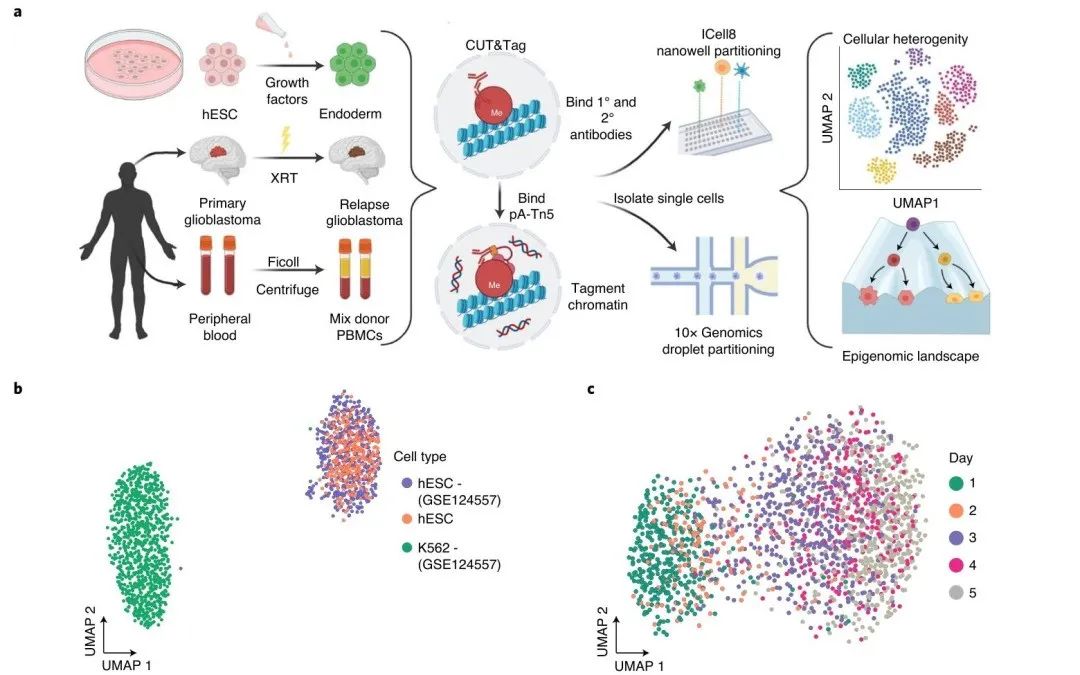
2.利用 scCUT&Tag 技术研究细胞分化过程中的基因沉默
实验设计与方法:研究人员将 hESCs 诱导分化为确定内胚层,通过对阶段特异性转录因子进行免疫荧光染色来确认分化情况(补充图 2a)。对 1,830 个单细胞的 H3K27me3 进行 scCUT&Tag 分析,每个细胞中位数为 279 个片段。
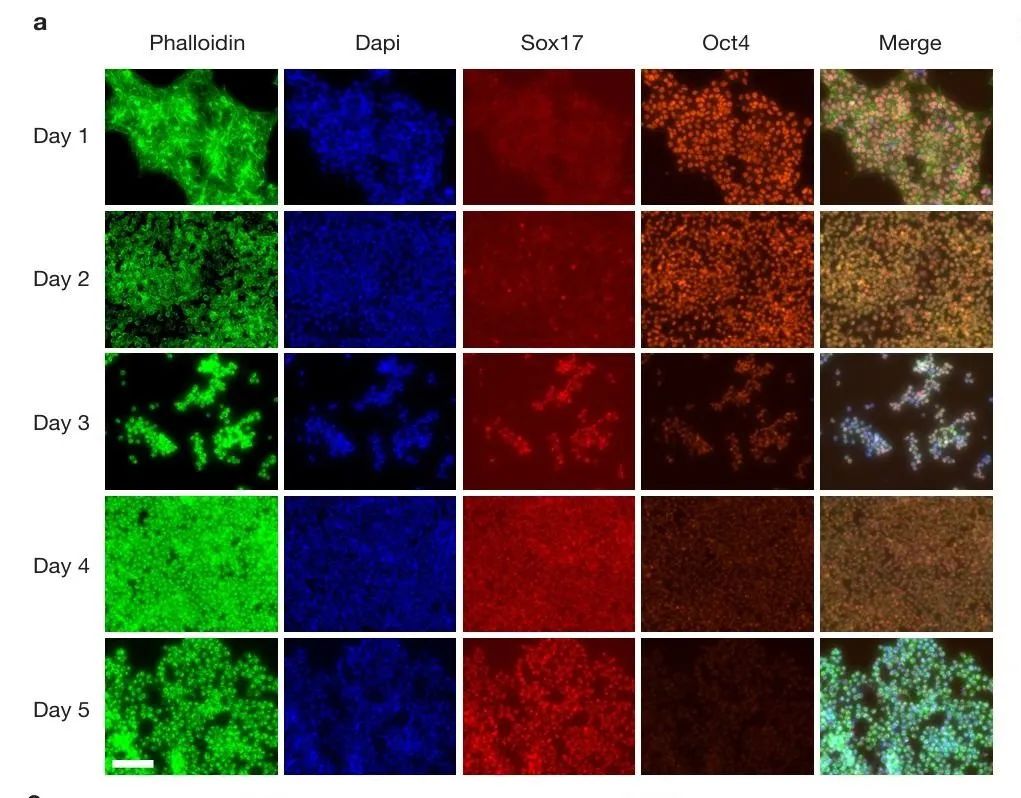
实验结果:UMAP 嵌入分析显示,从 hESCs 到确定内胚层存在一条清晰的发育轨迹(图 1c),且不受批次效应影响。在分化过程中,H3K27me3 信号与基因表达呈负相关。以干细胞和内胚层分化的已知标记基因为例,干细胞标记基因如 SOX2、KLF4 和 FOXD3 在 hESCs 中表达且缺乏 H3K27me3 修饰,但随着分化进程逐渐沉默;在分化第 2 - 3 天,细胞转变为中内胚层状态,中胚层标记基因的染色质沉默程度降低;随着分化继续,内胚层标记基因如 FOXA2、SOX17 和 PRDM1 被激活并失去 H3K27me3 信号(图 1d)。而外胚层标记基因 PAX6 和 LHX2 未表达且积累 H3K27me3,符合基因沉默特征。对单细胞进行伪时间排序也重现了上述实时结果。

3.scCUT&Tag 技术在复杂组织细胞类型分析中的应用
实验设计与方法:研究人员将 scCUT&Tag 技术应用于 10x Genomics 微流控平台,对来自两名健康供体的混合外周血单个核细胞(PBMCs)的 H3K27me3 进行分析。先对 100 万个细胞进行批量 scCUT&Tag 实验,然后将 10,000 个细胞核分别加载到 10x Genomics 微流控芯片的两个通道中,以获得技术重复(补充图 3)。通过计算染色质沉默分数(CSS),利用基因活性评分模型创建与给定基因座相关的总体信号代理。

补充图 3:scCUT&Tag 实验的重复分析。a) 小提琴图展示不同生物学和技术重复中细胞的独特片段数(中位数 3.03 - 3.09 ),数量相似。b)、c) UMAP 图分别按生物学、技术重复着色,显示样本重叠,表明供体和泳道对聚类影响小。
实验结果:经过质量控制筛选,得到 9,917 个细胞,每个细胞中位数为 1,110 个独特片段。通过 UMAP 嵌入分析和聚类,结合 CSS 评分以及公开的批量 H3K27me3 染色质免疫沉淀测序(ChIP - seq)数据投影,成功识别出 PBMCs 中的主要细胞类型,包括淋巴系(T 细胞、自然杀伤细胞和 B 细胞)和髓系(单核细胞)细胞(图 2a)。通过该方法,能够生成样本中不同细胞类型特异性的多梳蛋白组(PcG)景观,无需进行物理细胞分选,减少了批次效应、测序深度或样本异质性等干扰因素(补充图 3)。此外,研究人员还利用 souporcell 算法对每个生物供体的细胞进行解复用,能够区分不同供体的细胞,且聚类分析表明细胞聚类是由细胞类型差异驱动,而非供体特异性效应(图 2c)。

图 2:H3K27me3 的 scCUT&Tag 可轻松识别 PBMC 中的主要亚型
a:左图,PBMC 单细胞数据的 UMAP 嵌入图,无监督聚类揭示五个簇;右图,主要 PBMC 细胞类型的下采样 ChIP-seq 批量数据在单细胞 CUT&Tag 数据上的 UMAP 投影。
b:各簇中 H3K27me3 信号(CSS)显著低或高的基因热图,突出细胞类型特异性基因。
c:PBMC 数据基因型变异调用的稀疏混合模型聚类,按基因型分配染色(去除多重体之前)。
4.scCUT&Tag 技术在脑肿瘤研究中的应用
实验设计与方法:研究人员对 1,311 个来自原发性胶质母细胞瘤(手术切除后快速冷冻)的单细胞进行 H3K27me3 的 scCUT&Tag 分析,使用 10x scCUT&Tag 工作流程,每个细胞中位数为 3,643 个片段。通过构建 CSS 对先前定义的标记基因座进行注释,以识别不同细胞群。同时,将来自同一患者的胶质瘤干细胞系(UW7gsc)、两个已建立的神经干细胞系(U5 和 CB660)的 CUT&RUN 批量数据以及 ENCODE ChIP - seq 批量数据(单核细胞作为小胶质细胞的代理和星形胶质细胞)投影到 scCUT&Tag 肿瘤样本嵌入图上,以确认细胞群注释。
实验结果:在原发性胶质母细胞瘤样本中区分出四个主要细胞群(图 3a),分别注释为小胶质细胞(Cluster 1,PTPRC 基因处 CSS 较低)、神经元(Cluster 3,RBFOX3 基因处 CSS 较低)、少突胶质细胞(Cluster 4,MOBP 基因处 CSS 较低)和其他神经谱系细胞(包括肿瘤细胞,Cluster 4,SOX2 基因处 CSS 较低)(图 3b)。投影分析结果证实了 CSS 注释的准确性(图 3c)。

图 3:人类胶质母细胞瘤原发性和复发样本的 H3K27me3 的 scCUT&Tag 数据显示肿瘤细胞簇内 PcG 分布的异质性和治疗后的簇富集
a:基于 H3K27me3 信号的原发性人类胶质母细胞瘤单细胞的 UMAP 嵌入图。
b:使用关键标记基因的 CSS 进行簇注释,识别小胶质细胞、神经元、少突胶质细胞和肿瘤细胞。
c:批量 ChIP-seq 或批量 CUT&RUN 数据在患者样本上的 UMAP 变换和投影。
d:左图,原发性和复发样本肿瘤细胞的 UMAP 共嵌入图;右图,显示每个样本中属于各簇的细胞比例的条形图。
e:左图,两条以簇 T1 为起点,分别以簇 T4 或 T2 为终点的伪时间轨迹;右图,基于肿瘤细胞 ATAC-seq 数据中峰的 H3K27me3 活性的 132 个显著基序偏差的热图。
f:肿瘤细胞按选定基序偏差分数着色的 UMAP 图,展示不同阶段和轨迹特异性沉默的基序。
肿瘤治疗前后的变化分析:研究人员对患者手术后 5 个月复发样本(通过快速尸检获得)进行 H3K27me3 的 scCUT&Tag 分析,得到 1,168 个单细胞(每个细胞中位数为 16,232 个片段)。经过质量控制和低维嵌入分析,在复发样本中识别出四种不同细胞类型(补充图 6a)。
将复发样本的单细胞轮廓投影到原发性肿瘤的 UMAP 嵌入图上,鉴定出 71 个与肿瘤细胞簇共定位的细胞(补充图 6b)。通过共嵌入和分析原发性肿瘤和复发肿瘤细胞数据,发现复发肿瘤细胞中 Cluster T1 显著富集(图 3d)。

FRiP 分析表明,复发肿瘤细胞的背景信号高于原发性肿瘤细胞(补充图 6c)。基因集富集分析发现,Verhaak_glioblastoma_proneural 基因集在耐药细胞簇中沉默,而具有高 CpG 含量且在全脑中被 H3K27me3 标记的基因集在该肿瘤簇中缺乏 H3K27me3 信号,这表明胶质母细胞瘤细胞的 PcG 景观更类似于干细胞样状态,而非终末分化状态(补充图 7)。

补充图7:复发样本(集群一)基因集富集分析。对比集群一和其他集群基因,排序差异沉默基因并分析MSigDB基因集。a)正富集基因集含高PcG活性基因,Verhaak_glioblastoma_proneural基因集在列。b)负富集基因集含低PcG活性基因,以Meissner全脑H3K27me3相关基因集为代表,符合胶质母细胞瘤细胞特征。
细胞簇关系及转录因子程序差异分析:研究人员发现肿瘤细胞簇 T1、T2 和 T4 沿着一个连续体分布,而 Cluster T3 与主要肿瘤细胞群分离。通过单细胞 ATAC - seq 数据注释肿瘤细胞亚群中的增强子和启动子,基于 H3K27me3 信号计算转录因子(TF)基序富集和缺失情况。分析两条以 Cluster T1(假定为干细胞样簇)为起点,分别以 Cluster T4(轨迹 1)或 Cluster T2(轨迹 2)为终点的伪时间轨迹上的基序偏差(图 3e)。
结果发现,在轨迹顶点,存在一些共同沉默的基序,如 NEUROD1、SNAI2 和 TCF12 等 TF 的基序(图 3f 左列);在中间伪时间点,轨迹 1 有 NR1DA2 基序沉默,轨迹 2 有 ETV5 基序沉默,也有 RFX4 基序在两条轨迹中均沉默(图 3f 中列);随着伪时间推进,轨迹 1 出现 HES5 基序沉默,轨迹 2 出现 GATA6 基序沉默。此外,DNMT1 基序在两条伪时间终点均强烈沉默,这与 PcG 对 DNMT1 富集的启动子和增强子的沉默是分化的共同特征这一观点相符(图 3f 右列)。
四
scCUT&Tag技术为单细胞染色质修饰分析带来了突破性进展,然而,技术的发展永无止境,其仍存在可优化的空间。在实验流程方面,尽管该技术已实现单细胞层面的染色质修饰分析,但操作步骤相对繁琐,样本起始量要求以及实验成本等问题限制了其广泛应用。未来需要进一步简化流程,降低样本需求和成本,提高技术的普适性,让更多实验室能够轻松开展相关研究。
从数据分析角度来看,目前对scCUT&Tag数据的挖掘还不够深入。虽然已能通过数据区分细胞类型、解析分化过程和肿瘤异质性,但面对复杂的生物系统,现有的分析方法可能无法充分揭示染色质修饰与基因表达调控之间的深层次关系。因此,开发更精准、高效的数据分析算法和工具迫在眉睫,以深度解析海量数据,挖掘隐藏在其中的关键信息。
展望未来,scCUT&Tag技术有望与其他单细胞技术,如单细胞RNA测序、单细胞蛋白质组学等进行多组学整合。这种整合能够从不同层面全面描绘细胞的状态,为理解细胞命运决定、疾病发生发展机制提供更完整的视角。同时,随着技术的不断完善,scCUT&Tag在临床诊断、药物研发等领域的应用潜力将进一步释放,为精准医学的发展提供强大助力,推动生命科学研究迈向新的高度。
(点击阅读原文获取文献)

